A génexpressziós adatok vizualizálásának és értelmezésének számos módszere használható mind a microarray, mind az RNS-seq kísérletekhez. Az alábbiakban a leggyakoribb módszerek közül néhányat tárgyalunk.
Hőtérképek és klaszterezés
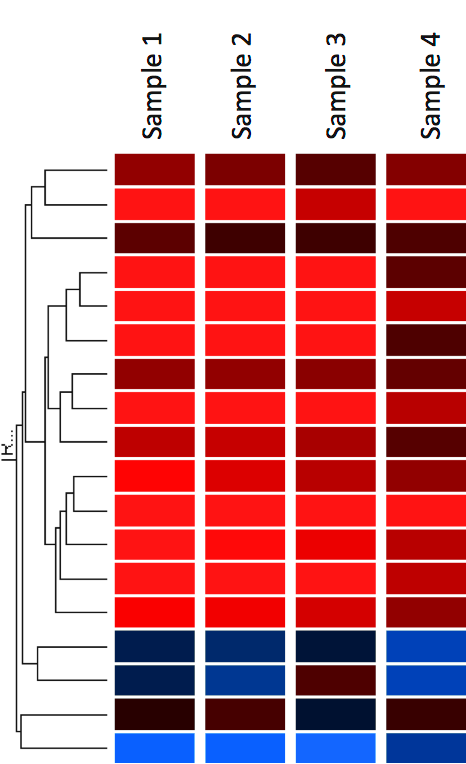
A génexpressziós adatok vizualizálásának gyakori módszere a hőtérképként való megjelenítés (12. ábra). A hőtérkép kombinálható klaszterezési módszerekkel is, amelyek a gének és/vagy minták génexpressziós mintázatának hasonlósága alapján csoportosítják őket. Ez hasznos lehet a közösen szabályozott gének vagy egy adott állapothoz (pl. betegséghez vagy környezeti feltételhez) kapcsolódó biológiai szignatúrák azonosítására (4).
A hőtérképeken az adatokat egy rácshálóban jelenítik meg, ahol minden sor egy gént, minden oszlop pedig egy mintát jelöl. A dobozok színe és intenzitása a génexpresszió változásainak (nem abszolút értékeinek) ábrázolására szolgál. Az alábbi példában a piros szín a felfelé szabályozott géneket, a kék pedig a lefelé szabályozott géneket jelöli. A fekete változatlan expressziót jelent.
Génkészlet-gazdagodási elemzés és útvonalelemzés
A génexpressziós adatok értelmezésének gyakori megközelítése a génkészlet-gazdagodási elemzés, amely a differenciálisan kifejezett gének funkcionális annotációján alapul (13. ábra). Ez hasznos annak megállapítására, hogy a differenciálisan expresszálódó gének kapcsolatban állnak-e egy bizonyos biológiai folyamattal vagy molekuláris funkcióval.
A géntermékek szabványosított annotációját tartalmazó Gene Ontologyt gyakran használják erre a célra. Ez úgy működik, hogy a génlistában szereplő egyes annotációk (pl. differenciálisan expresszált gének) gyakoriságát összehasonlítja egy referencialistával (általában a microarray-n vagy a genomban lévő összes génnel). A KEGG, az Ingenuity, a Reactome vagy a WikiPathways által szolgáltatott biológiai útvonalak gazdagítása hasonló módon végezhető (12,13).
A génkészlet-dúsítás és az útvonalelemzés népszerű eszközei közé tartoznak a következők:
- DAVID (ingyenes online eszköz)
- GSEA (ingyenes)
- Ingenuity (licencköteles)
- Reactome (ingyenes)

Hálózatelemzés
A hálózatelemzés kiegészíti az útvonalelemzést, és arra használható, hogy megmutassa, hogyan hatnak egymásra a különböző útvonalak kulcskomponensei. Ez hasznos lehet a több biológiai folyamatot és útvonalat befolyásoló szabályozási események azonosításához (12,13).