Muitos dos métodos de visualização e interpretação de dados de expressão gênica podem ser usados tanto para experimentos de microarranjo quanto para RNA-seq. Alguns dos métodos mais comuns são discutidos abaixo.
Heatmaps and clustering
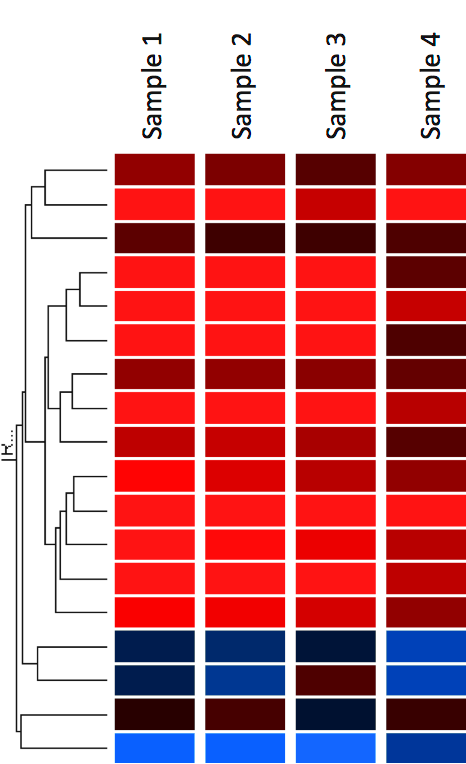
Um método comum de visualização de dados de expressão gênica é mostrá-los como um mapa térmico (Figura 12). O heatmap também pode ser combinado com métodos de agrupamento que agrupam genes e/ou amostras com base na similaridade do seu padrão de expressão gênica. Isto pode ser útil para identificar genes que são normalmente regulados, ou assinaturas biológicas associadas a uma condição particular (por exemplo, uma doença ou uma condição ambiental) (4).
Nos mapas de calor os dados são mostrados numa grelha onde cada linha representa um gene e cada coluna representa uma amostra. A cor e intensidade das caixas é usada para representar as mudanças (não valores absolutos) da expressão gênica. No exemplo abaixo, vermelho representa genes regulados para cima e azul representa genes regulados para baixo. Preto representa a expressão inalterada.
Análise de enriquecimento do conjunto genético e análise de caminho
Uma abordagem comum para interpretar os dados de expressão gênica é a análise de enriquecimento do conjunto genético com base na anotação funcional dos genes diferencialmente expressos (Figura 13). Isto é útil para descobrir se os genes diferentemente expressos estão associados a um determinado processo biológico ou função molecular.
A Ontologia Genética, contendo anotação padronizada de produtos gênicos, é comumente usada para este propósito. Ela funciona comparando a frequência de anotações individuais na lista de genes (por exemplo, genes diferentemente expressos) com uma lista de referência (geralmente todos os genes no microarranjo ou no genoma). O enriquecimento das vias biológicas fornecidas pelo KEGG, Ingenuidade, Reactoma ou WikiPathways pode ser realizado de forma semelhante (12,13).
Ferramentas populares para o enriquecimento do conjunto genético e análise das vias incluem:
- DAVID (ferramenta online gratuita)
- GSEA (livre)
- Ingenuidade (licença requerida)
- Reactome (livre)

Análise da rede
Análise da rede é complementar à análise do caminho e pode ser usada para mostrar como componentes chave de diferentes caminhos interagem. Isto pode ser útil para identificar eventos regulatórios que influenciam múltiplos processos e caminhos biológicos (12,13).