Takeaways
-
Ein „Deep Learning“-Softwareprogramm des zu Google gehörenden Labors DeepMind zeigte große Fortschritte bei der Lösung einer der größten Herausforderungen der Biologie – dem Verständnis der Proteinfaltung.
-
Die Proteinfaltung ist der Prozess, durch den ein Protein seine Form von einer Reihe von Bausteinen zu seiner endgültigen dreidimensionalen Struktur annimmt, die seine Funktion bestimmt.
-
Wenn Wissenschaftler besser vorhersagen können, wie Proteine ihre Struktur annehmen oder sich „falten“, können sie schneller Medikamente entwickeln, die zum Beispiel die Wirkung wichtiger viraler Proteine blockieren.
Die Lösung dessen, was Biologen das „Proteinfaltungsproblem“ nennen, ist eine große Sache. Proteine sind die Arbeitspferde der Zellen und kommen in allen lebenden Organismen vor. Sie bestehen aus langen Ketten von Aminosäuren und sind für die Struktur der Zellen und die Kommunikation zwischen ihnen sowie für die Regulierung der gesamten Chemie im Körper unerlässlich.
Das Google-eigene Unternehmen für künstliche Intelligenz DeepMind hat in dieser Woche ein Deep-Learning-Programm namens AlphaFold2 vorgestellt, das von Experten als Durchbruch bei der Lösung der großen Herausforderung der Proteinfaltung bezeichnet wird.
Proteine sind lange Ketten von Aminosäuren, die wie Perlen auf einer Schnur miteinander verbunden sind. Doch damit ein Protein seine Aufgabe in der Zelle erfüllen kann, muss es sich „falten“ – ein Prozess des Verdrehens und Biegens, der das Molekül in eine komplexe dreidimensionale Struktur verwandelt, die mit seinem Ziel in der Zelle interagieren kann. Wenn die Faltung gestört ist, nimmt das Protein nicht die richtige Form an – und kann seine Aufgabe im Körper nicht erfüllen. Dies kann zu Krankheiten führen, wie es bei häufigen Krankheiten wie Alzheimer und seltenen Krankheiten wie Mukoviszidose der Fall ist.
Deep Learning ist eine Computertechnik, die die oft verborgenen Informationen in großen Datensätzen nutzt, um interessante Fragen zu lösen. Sie wird in Bereichen wie Spiele, Sprach- und Stimmerkennung, autonome Autos, Wissenschaft und Medizin eingesetzt.
Ich glaube, dass Werkzeuge wie AlphaFold2 den Wissenschaftlern dabei helfen werden, neue Arten von Proteinen zu entwickeln, die zum Beispiel dabei helfen können, Kunststoffe abzubauen und künftige Virusepidemien und Krankheiten zu bekämpfen.
Ich bin Computerchemikerin und Autorin des Buches The State of Science. Meine Studenten und ich untersuchen die Struktur und die Eigenschaften von fluoreszierenden Proteinen mit Hilfe von Computerprogrammen zur Proteinfaltung, die auf der klassischen Physik basieren.
Nach jahrzehntelangem Studium durch Tausende von Forschergruppen sind diese Programme zur Vorhersage der Proteinfaltung sehr gut in der Lage, strukturelle Veränderungen zu berechnen, die auftreten, wenn wir kleine Änderungen an bekannten Molekülen vornehmen.
Aber sie sind nicht in der Lage, die Faltung von Proteinen von Grund auf vorherzusagen. Bevor Deep Learning aufkam, schien das Problem der Proteinfaltung unlösbar zu sein, und es war absehbar, dass es die Computerchemiker noch viele Jahrzehnte lang frustrieren würde.
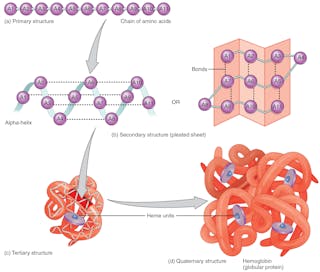
Proteinfaltung
Die Sequenz der Aminosäuren – die in der DNA verschlüsselt ist – definiert die 3D-Form des Proteins. Die Form bestimmt seine Funktion. Wenn sich die Struktur des Proteins ändert, kann es seine Funktion nicht mehr erfüllen. Die korrekte Vorhersage von Proteinfalten auf der Grundlage der Aminosäuresequenz könnte die Entwicklung von Medikamenten revolutionieren und die Ursachen neuer und alter Krankheiten erklären.
Alle Proteine mit derselben Sequenz von Aminosäurebausteinen falten sich in dieselbe dreidimensionale Form, wodurch die Wechselwirkungen zwischen den Aminosäuren optimiert werden. Sie tun dies innerhalb von Millisekunden, obwohl ihnen eine astronomische Anzahl von möglichen Konfigurationen zur Verfügung steht – etwa 10 hoch 300. Diese enorme Anzahl macht es schwierig, die Faltung eines Proteins vorherzusagen, selbst wenn die Wissenschaftler die vollständige Sequenz der Aminosäuren kennen, aus denen das Protein besteht. Früher war es unmöglich, die Struktur eines Proteins anhand der Aminosäuresequenz vorherzusagen. Proteinstrukturen mussten experimentell bestimmt werden, ein zeitaufwändiges und teures Unterfangen.
Wenn Forscher besser vorhersagen können, wie sich Proteine falten, können sie besser verstehen, wie Zellen funktionieren und wie falsch gefaltete Proteine Krankheiten verursachen. Bessere Werkzeuge zur Vorhersage von Proteinen werden uns auch dabei helfen, Medikamente zu entwickeln, die auf eine bestimmte topologische Region eines Proteins abzielen, in der chemische Reaktionen ablaufen.

AlphaFold ist aus Deep-Learning-Schach-, Go- und Pokerspielen hervorgegangen
Der Erfolg des DeepMind-Programms zur Vorhersage der Proteinfaltung, AlphaFold genannt, kommt nicht unerwartet. Andere von DeepMind geschriebene Deep-Learning-Programme haben die weltbesten Schach-, Go- und Pokerspieler in die Knie gezwungen.
Im Jahr 2016 war Stockfish-8, eine Open-Source-Schachengine, der Weltmeister im Computerschach. Sie bewertete 70 Millionen Schachpositionen pro Sekunde und konnte auf jahrhundertelang gesammelte menschliche Schachstrategien und jahrzehntelange Computererfahrung zurückgreifen. Sie spielte effizient und brutal und schlug alle ihre menschlichen Herausforderer gnadenlos und ohne einen Hauch von Finesse. Jetzt kommt Deep Learning ins Spiel.
Am 7. Dezember 2017 besiegte das Deep-Learning-Schachprogramm AlphaZero von Google Stockfish-8. Die Schachengines spielten 100 Partien, wobei AlphaZero 28 gewann und 72 unentschieden spielte. Es hat kein einziges Spiel verloren. AlphaZero führte nur 80.000 Berechnungen pro Sekunde durch, im Gegensatz zu den 70 Millionen Berechnungen von Stockfish-8, und es brauchte nur vier Stunden, um Schach von Grund auf zu lernen, indem es einige Millionen Mal gegen sich selbst spielte und seine neuronalen Netzwerke optimierte, während es aus seinen Erfahrungen lernte.
AlphaZero hat nichts von Menschen oder von Menschen gespielten Schachpartien gelernt. Es hat sich selbst etwas beigebracht und dabei Strategien entwickelt, die es nie zuvor gesehen hat. In einem Kommentar in der Zeitschrift Science schrieb der ehemalige Schachweltmeister Garry Kasparov, dass AlphaZero durch das Lernen aus dem eigenen Spiel Strategien entwickelte, die „die Wahrheit“ des Schachs widerspiegeln und nicht „die Prioritäten und Vorurteile“ der Programmierer. „Es ist die Verkörperung des Klischees ‚work smarter, not harder‘.“
CASP – die Olympiade für Molekularmodellierer
Alle zwei Jahre testen die weltbesten Computerchemiker die Fähigkeiten ihrer Programme zur Vorhersage der Faltung von Proteinen und treten im Wettbewerb Critical Assessment of Structure Prediction (CASP) gegeneinander an.
Bei diesem Wettbewerb erhalten die Teams die lineare Aminosäuresequenz von etwa 100 Proteinen, deren 3D-Form zwar bekannt ist, aber noch nicht veröffentlicht wurde, und müssen berechnen, wie sich diese Sequenzen falten würden. Im Jahr 2018 schlug AlphaFold, der Deep-Learning-Neuling im Wettbewerb, alle traditionellen Programme – aber nur knapp.
Zwei Jahre später, am Montag, wurde bekannt gegeben, dass Alphafold2 den Wettbewerb 2020 mit großem Vorsprung gewonnen hat. Es schlug seine Konkurrenten, und seine Vorhersagen waren vergleichbar mit den vorhandenen experimentellen Ergebnissen, die mit Goldstandardtechniken wie Röntgenbeugungskristallographie und Kryo-Elektronenmikroskopie ermittelt wurden. Ich gehe davon aus, dass AlphaFold2 und seine Nachkommen bald die Methode der Wahl sein werden, um Proteinstrukturen zu bestimmen, bevor man auf experimentelle Techniken zurückgreift, die mühsame, aufwändige Arbeit an teuren Instrumenten erfordern.
Einer der Gründe für den Erfolg von AlphaFold2 ist, dass es die Protein-Datenbank, die über 170.000 experimentell bestimmte 3D-Strukturen enthält, nutzen konnte, um sich selbst zu trainieren, um die korrekt gefalteten Strukturen von Proteinen zu berechnen.
Die potenzielle Bedeutung von AlphaFold lässt sich ermessen, wenn man die Zahl aller veröffentlichten Proteinstrukturen – etwa 170.000 – mit den 180 Millionen DNA- und Proteinsequenzen vergleicht, die in der Universal Protein Database hinterlegt sind. AlphaFold wird uns helfen, die Schatzkammern von DNA-Sequenzen zu durchforsten und nach neuen Proteinen mit einzigartigen Strukturen und Funktionen zu suchen.
Hat AlphaFold mich, einen Molekularmodellierer, überflüssig gemacht?
Wie bei den Schach- und Go-Programmen – AlphaZero und AlphaGo – wissen wir nicht genau, was der AlphaFold2-Algorithmus tut und warum er bestimmte Korrelationen verwendet, aber wir wissen, dass er funktioniert.
Das Verständnis der „Denkweise“ von AlphaFold wird uns nicht nur dabei helfen, die Strukturen wichtiger Proteine vorherzusagen, sondern auch neue Erkenntnisse über den Mechanismus der Proteinfaltung zu gewinnen.
Eine der häufigsten Befürchtungen, die in Bezug auf KI geäußert werden, ist, dass sie zu Massenarbeitslosigkeit führen wird. AlphaFold hat noch einen weiten Weg vor sich, bevor es die Proteinfaltung konsistent und erfolgreich vorhersagen kann.
Wenn es jedoch einmal ausgereift ist und das Programm die Proteinfaltung simulieren kann, werden Computerchemiker maßgeblich an der Verbesserung der Programme beteiligt sein und versuchen, die zugrunde liegenden Zusammenhänge zu verstehen und das Programm zur Lösung wichtiger Probleme wie der Fehlfaltung von Proteinen im Zusammenhang mit vielen Krankheiten wie Alzheimer, Parkinson, Mukoviszidose und Chorea Huntington einzusetzen.
AlphaFold und seine Nachkommen werden sicherlich die Arbeitsweise der Computerchemiker verändern, aber sie nicht überflüssig machen. Andere Bereiche werden nicht so viel Glück haben. In der Vergangenheit konnten Roboter Menschen bei manueller Arbeit ersetzen; mit der KI werden auch unsere kognitiven Fähigkeiten in Frage gestellt.