Takeaways
-
Un programma software di “deep learning” del laboratorio DeepMind, di proprietà di Google, ha mostrato grandi progressi nel risolvere una delle più grandi sfide della biologia – capire la piegatura delle proteine.
-
Il ripiegamento delle proteine è il processo con cui una proteina prende la sua forma da una stringa di blocchi di costruzione alla sua struttura tridimensionale finale, che determina la sua funzione.
-
Per prevedere meglio come le proteine prendono la loro struttura, o “ripiegamento”, gli scienziati possono sviluppare più rapidamente farmaci che, per esempio, bloccano l’azione di proteine virali cruciali.
Solvere quello che i biologi chiamano “il problema del ripiegamento delle proteine” è un grosso problema. Le proteine sono i cavalli di battaglia delle cellule e sono presenti in tutti gli organismi viventi. Sono costituite da lunghe catene di aminoacidi e sono vitali per la struttura delle cellule e la comunicazione tra di esse, oltre a regolare tutta la chimica del corpo.
Questa settimana, la società di intelligenza artificiale DeepMind, di proprietà di Google, ha dimostrato un programma di apprendimento profondo chiamato AlphaFold2, che gli esperti chiamano una svolta verso la soluzione della grande sfida del ripiegamento delle proteine.
Le proteine sono lunghe catene di aminoacidi collegate insieme come perline su una corda. Ma perché una proteina faccia il suo lavoro nella cellula, deve “piegarsi” – un processo di torsione e piegatura che trasforma la molecola in una complessa struttura tridimensionale che può interagire con il suo bersaglio nella cellula. Se il ripiegamento è interrotto, allora la proteina non formerà la forma corretta – e non sarà in grado di svolgere il suo lavoro all’interno del corpo. Questo può portare alla malattia – come nel caso di una malattia comune come l’Alzheimer, e di quelle rare come la fibrosi cistica.
Il deep learning è una tecnica computazionale che utilizza le informazioni spesso nascoste contenute in vaste serie di dati per risolvere questioni di interesse. È stata ampiamente utilizzata in campi come i giochi, il riconoscimento vocale e del parlato, le auto autonome, la scienza e la medicina.
Credo che strumenti come AlphaFold2 aiuteranno gli scienziati a progettare nuovi tipi di proteine, quelle che potrebbero, per esempio, aiutare a scomporre la plastica e combattere future pandemie virali e malattie.
Sono un chimico computazionale e autore del libro The State of Science. I miei studenti ed io studiamo la struttura e le proprietà delle proteine fluorescenti usando programmi informatici di ripiegamento delle proteine basati sulla fisica classica.
Dopo decenni di studio da parte di migliaia di gruppi di ricerca, questi programmi di previsione del ripiegamento delle proteine sono molto bravi a calcolare i cambiamenti strutturali che si verificano quando apportiamo piccole alterazioni a molecole note.
Ma non sono riusciti a prevedere adeguatamente come le proteine si piegano da zero. Prima che arrivasse il deep learning, il problema del ripiegamento delle proteine sembrava impossibilmente difficile, e sembrava destinato a frustrare i chimici computazionali per molti decenni a venire.
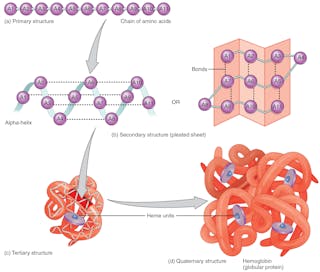
Piegamento delle proteine
La sequenza degli amminoacidi – che è codificata nel DNA – definisce la forma 3D della proteina. La forma determina la sua funzione. Se la struttura della proteina cambia, essa non è in grado di svolgere la sua funzione. Prevedere correttamente le pieghe delle proteine in base alla sequenza degli amminoacidi potrebbe rivoluzionare il design dei farmaci e spiegare le cause di nuove e vecchie malattie.
Tutte le proteine con la stessa sequenza di amminoacidi si piegano nella stessa forma tridimensionale, che ottimizza le interazioni tra gli amminoacidi. Lo fanno in pochi millisecondi, anche se hanno un numero astronomico di possibili configurazioni a loro disposizione – circa 10 alla potenza di 300. Questo numero enorme è ciò che rende difficile prevedere come si ripiega una proteina anche quando gli scienziati conoscono la sequenza completa degli aminoacidi che la compongono. In precedenza, prevedere la struttura delle proteine dalla sequenza degli aminoacidi era impossibile. Le strutture delle proteine venivano determinate sperimentalmente, un’impresa lunga e costosa.
Una volta che i ricercatori potranno prevedere meglio come si piegano le proteine, saranno in grado di capire meglio come funzionano le cellule e come le proteine mal piegate causano le malattie. Migliori strumenti di predizione delle proteine ci aiuteranno anche a progettare farmaci che possono prendere di mira una particolare regione topologica di una proteina dove avvengono le reazioni chimiche.

AlphaFold nasce dall’apprendimento profondo delle partite di scacchi, Go e poker
Il successo del programma di previsione del ripiegamento delle proteine di DeepMind, chiamato AlphaFold, non è inaspettato. Altri programmi di deep-learning scritti da DeepMind hanno demolito i migliori giocatori di scacchi, Go e poker del mondo.
Nel 2016 Stockfish-8, un motore di scacchi open-source, è stato il campione mondiale di scacchi al computer. Valutava 70 milioni di posizioni di scacchi al secondo e aveva secoli di strategie scacchistiche umane accumulate e decenni di esperienza informatica da cui attingere. Ha giocato in modo efficiente e brutale, battendo senza pietà tutti i suoi sfidanti umani senza un grammo di finezza. Inserisci l’apprendimento profondo.
Il 7 dicembre 2017, il programma di scacchi ad apprendimento profondo di Google AlphaZero ha battuto Stockfish-8. I motori di scacchi hanno giocato 100 partite, con AlphaZero che ha vinto 28 e pareggiato 72. Non ha perso una sola partita. AlphaZero ha fatto solo 80.000 calcoli al secondo, contro i 70 milioni di Stockfish-8, e ha impiegato solo quattro ore per imparare gli scacchi da zero, giocando contro se stesso alcuni milioni di volte e ottimizzando le sue reti neurali mentre imparava dalla sua esperienza.
AlphaZero non ha imparato nulla dagli umani o dalle partite di scacchi giocate dagli umani. Ha imparato da solo e, nel processo, ha derivato strategie mai viste prima. In un commento sulla rivista Science, l’ex campione del mondo di scacchi Garry Kasparov ha scritto che imparando dal gioco stesso, AlphaZero ha sviluppato strategie che “riflettono la verità” degli scacchi piuttosto che riflettere “le priorità e i pregiudizi” dei programmatori. “È l’incarnazione del cliché ‘lavora in modo più intelligente, non più difficile'”.
CASP – le Olimpiadi dei modellatori molecolari
Ogni due anni, i migliori chimici computazionali del mondo mettono alla prova le capacità dei loro programmi di prevedere il ripiegamento delle proteine e competono nella competizione Critical Assessment of Structure Prediction (CASP).
Nella competizione, le squadre ricevono la sequenza lineare di aminoacidi per circa 100 proteine di cui è nota la forma 3D ma che non è ancora stata pubblicata; devono poi calcolare come queste sequenze si piegherebbero. Nel 2018 AlphaFold, l’esordiente deep-learning alla competizione, ha battuto tutti i programmi tradizionali – ma a malapena.
Due anni dopo, lunedì, è stato annunciato che Alphafold2 ha vinto la competizione del 2020 con un buon margine. Ha sbaragliato i suoi concorrenti, e le sue previsioni erano paragonabili ai risultati sperimentali esistenti determinati attraverso tecniche gold standard come la cristallografia a diffrazione di raggi X e la microscopia crioelettronica. Presto mi aspetto che AlphaFold2 e la sua progenie saranno i metodi di scelta per determinare le strutture delle proteine prima di ricorrere a tecniche sperimentali che richiedono un lavoro minuzioso e laborioso su strumentazione costosa.
Una delle ragioni del successo di AlphaFold2 è che potrebbe usare il database delle proteine, che ha oltre 170.000 strutture 3D determinate sperimentalmente, per allenarsi a calcolare le strutture correttamente piegate delle proteine.
Il potenziale impatto di AlphaFold può essere apprezzato se si confronta il numero di tutte le strutture proteiche pubblicate – circa 170.000 – con i 180 milioni di sequenze di DNA e proteine depositate nell’Universal Protein Database. AlphaFold ci aiuterà a smistare un tesoro di sequenze di DNA alla ricerca di nuove proteine con strutture e funzioni uniche.
Ha reso me, un modellatore molecolare, ridondante?
Come per i programmi di scacchi e Go – AlphaZero e AlphaGo – non sappiamo esattamente cosa faccia l’algoritmo AlphaFold2 e perché usi certe correlazioni, ma sappiamo che funziona.
Oltre ad aiutarci a prevedere le strutture di importanti proteine, la comprensione del “pensiero” di AlphaFold ci aiuterà anche a ottenere nuove intuizioni sul meccanismo di ripiegamento delle proteine.
Una delle paure più comuni espresse sull’IA è che porterà alla disoccupazione su larga scala. AlphaFold ha ancora molta strada da fare prima di riuscire a prevedere in modo coerente e con successo il ripiegamento delle proteine.
Tuttavia, una volta che sarà maturato e il programma potrà simulare il ripiegamento delle proteine, i chimici computazionali saranno integralmente coinvolti nel migliorare i programmi, cercando di capire le correlazioni sottostanti utilizzate, e applicando il programma per risolvere problemi importanti come il misfolding delle proteine associato a molte malattie come l’Alzheimer, il Parkinson, la fibrosi cistica e la malattia di Huntington.
AlphaFold e la sua progenie cambieranno certamente il modo di lavorare dei chimici computazionali, ma non li renderà ridondanti. Altri settori non saranno così fortunati. In passato i robot sono stati in grado di sostituire gli esseri umani che facevano lavori manuali; con l’IA, anche le nostre capacità cognitive vengono messe alla prova.