Många av metoderna för att visualisera och tolka genuttrycksdata kan användas för både mikroarray- och RNA-seq-experiment. Några av de vanligaste metoderna diskuteras nedan.
Heatmaps och klusterindelning
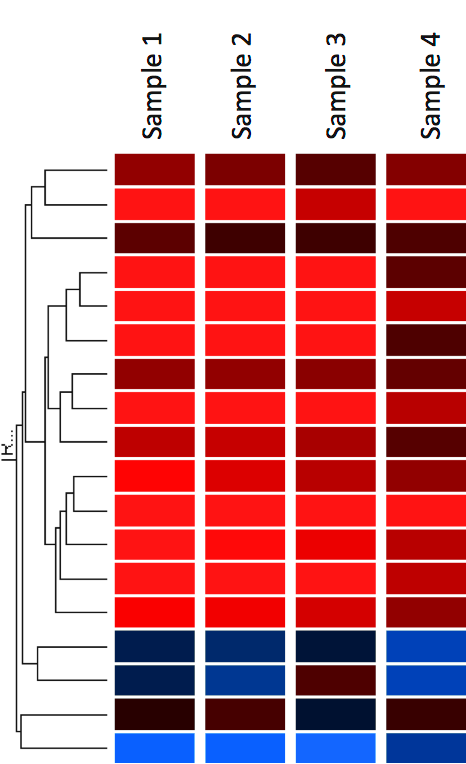
En vanlig metod för att visualisera genuttrycksdata är att visa dem som en heatmap (figur 12). Värmekartan kan också kombineras med klustermetoder som grupperar gener och/eller prover tillsammans baserat på likheten i deras genuttrycksmönster. Detta kan vara användbart för att identifiera gener som är gemensamt reglerade, eller biologiska signaturer som är förknippade med ett visst tillstånd (t.ex. en sjukdom eller ett miljötillstånd) (4).
I värmekartor visas data i ett rutnät där varje rad representerar en gen och varje kolumn representerar ett prov. Färgen och intensiteten i rutorna används för att representera förändringar (inte absoluta värden) av genuttrycket. I exemplet nedan representerar rött uppreglerade gener och blått nedreglerade gener. Svart representerar oförändrat uttryck.
Gen set enrichment analysis and pathway analysis
En vanlig metod för att tolka genuttrycksdata är gen set enrichment analysis baserad på funktionell annotering av de differentiellt uttryckta generna (figur 13). Detta är användbart för att ta reda på om de differentiellt uttryckta generna är associerade med en viss biologisk process eller molekylär funktion.
Gene Ontology, som innehåller standardiserad annotering av genprodukter, används vanligen för detta ändamål. Den fungerar genom att jämföra frekvensen av enskilda annotationer i genlistan (t.ex. differentiellt uttryckta gener) med en referenslista (vanligen alla gener på mikroarrayet eller i genomet). Anrikning av biologiska vägar som tillhandahålls av KEGG, Ingenuity, Reactome eller WikiPathways kan utföras på ett liknande sätt (12,13).
Populära verktyg för anrikning av genuppsättningar och analys av vägar inkluderar:
- DAVID (gratis online-verktyg)
- GSEA (gratis)
- Ingenuity (licens krävs)
- Reactome (gratis)

Nätverksanalys
Nätverksanalys är ett komplement till analys av vägar och kan användas för att visa hur nyckelkomponenter i olika vägar interagerar. Detta kan vara användbart för att identifiera regulatoriska händelser som påverkar flera biologiska processer och vägar (12,13).